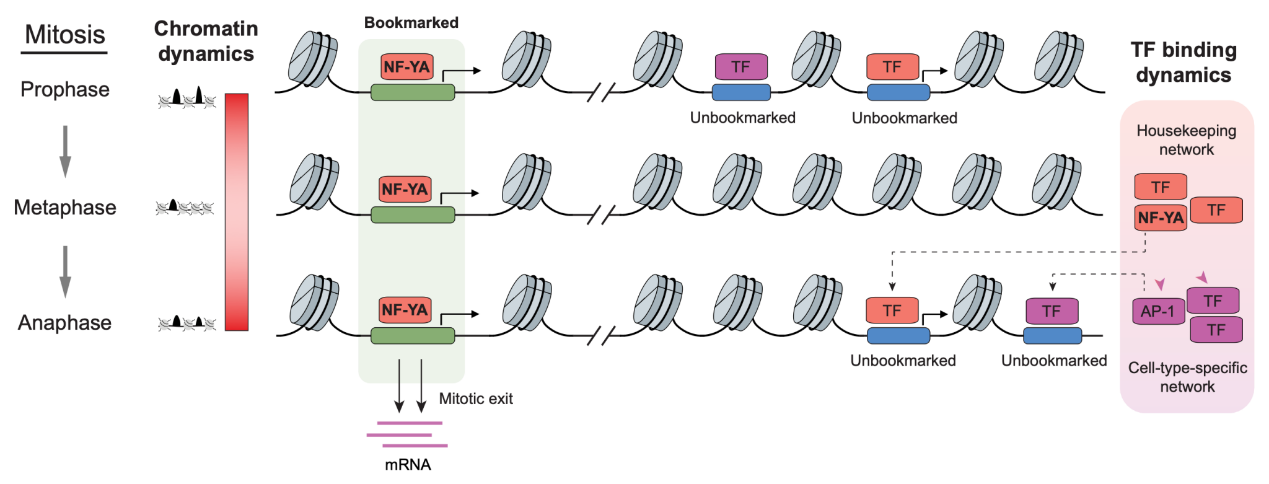
1、揭示细胞有丝分裂期转录调控动态机制(Science Advances,2023 Jan 25;9(4):eadd2175)
中国科学技术大学生命科学与医学部瞿昆教授课题组和合肥微尺度物质科学国家研究中心王志凯团队合作在细胞有丝分裂转录调控方面取得重要成果。相关成果以“Dynamics and regulation of mitotic chromatin accessibility bookmarking at single-cell resolution”为题发表于Science Advances杂志。在该研究中,团队绘制了来自有丝分裂不同时期的6,538个L02胎肝细胞的高质量染色质可及性图谱,并通过假时间轨迹、多组学整合、原位成像、转录因子扰动等干湿结合的分析策略系统性探究了有丝分裂期染色质状态和转录调控的动态变化。研究揭示了有丝分裂期细胞染色质可及性先减少后增加的动态变化和作为”转录记忆”载体始终活跃的染色质可及区域,发现了大规模转录因子与染色质的动态相互作用过程,从中鉴定了新的调控基因重激活的转录因子NF-YA,从而构建整个有丝分裂期转录调控网络的动态演变过程。
2、揭示肝转移癌免疫逃逸新机制及靶向治疗策略(J Clin Invest. 2023 Apr 17;133(8):e157937)
中国科学技术大学生命科学与医学部曾筑天课题组在国际知名医学期刊《Journal of Clinical Investigation》上在线发表了题为“In situ expansion and reprogramming of Kupffer cells elicits potent tumoricidal immunity against liver metastasis”的研究论文,该研究揭示了肝转移癌逃逸肝脏驻留巨噬细胞(Kupffer cells, KC)免疫监视的新机制,相应开发了原位靶向扩增并重塑KC抗肿瘤功能的新方法,在多种终末期肝转移癌动物模型中实现了肿瘤清除,从而为临床上转移性肝癌的免疫治疗策略开发提供了新思路。

3、发现自然杀伤细胞失去抗肿瘤功能关键机制(Nature Immunology volume 24, pages802–813(2023))
中国科学技术大学生命科学与医学部魏海明教授、郑小虎教授和田志刚教授课题组,与中国科学技术大学化学与材料科学学院黄光明课题组以及安徽医科大学第一附属医院钱叶本主任合作,发现肿瘤组织微环境NK细胞丢失表面膜突起,无法识别肿瘤细胞,失去了抗肿瘤功能。他们还创建了一种“单个免疫细胞膜质谱检测技术”,揭示NK细胞膜主要组分鞘磷脂的丢失是NK细胞丢失表面突起主要原因。研究成果2023年3月23日以“Tumors evade immune cytotoxicity by altering the surface topology of NK cells”为题,在线发表于《自然·免疫》(Nature Immunology)杂志,本研究揭示了一种肿瘤免疫逃逸的新机制,为基于NK细胞的肿瘤免疫治疗提供了新思路与新靶标。

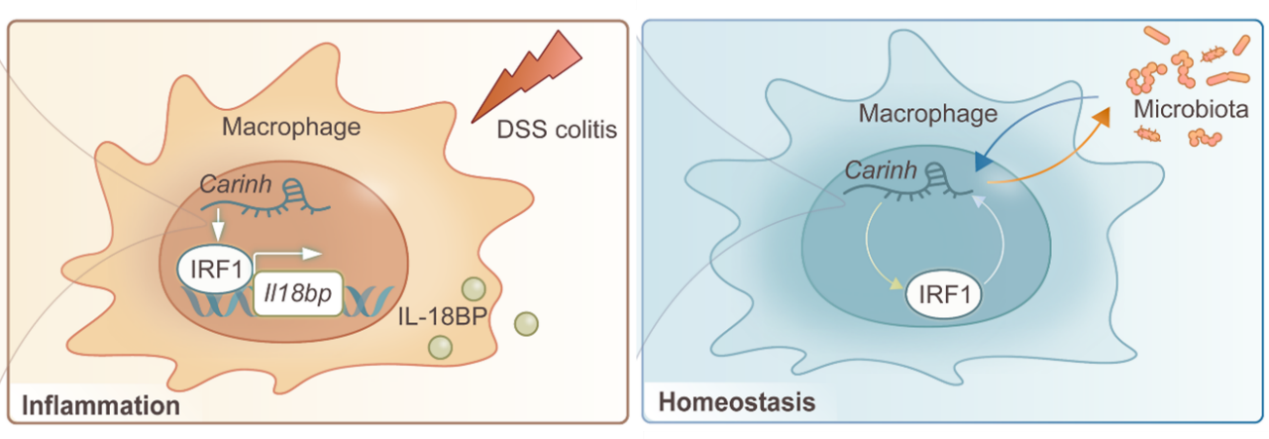
4、发现炎症性肠病(IBD)的新型易感基因及其致病机制(Cell Research volume 33, pages372–388 (2023))
炎症性肠病(Inflammatory bowel disease, IBD)是一种受到多因素影响的慢性炎症性肠道疾病,目前临床治疗手段有局限性,存在药物应答率低,以及耐药性等问题。针对这些临床治疗难题,筛选该疾病的新型敏感标志物,并明确其致病机制与关键致病因子,建立临床靶向阻断关键致病因子的新型个体化诊疗策略有助于提高药物应答率,真正做到“对症下药”。中国科学技术大学生命科学与医学部朱书教授团队发现IBD新型易感基因Carinh,并揭示该基因通过影响肠道菌群的组成和肠道免疫微环境,参与了IBD的发生发展。在机制研究中,发现Carinh通过调控肠炎中重要的炎症抑制因子IL-18BP的表达,减轻肠道中最为丰富的炎性因子IL-18介导的炎症应答,从而减缓IBD的发展。研究成果以《A lncRNA from an inflammatory bowel disease risk locus maintains intestinal host-commensal homeostasis》为题于2023年4月13日在线发表于《细胞研究》(Cell Research)杂志,该研究鉴定了IBD新型易感基因Carinh可以作为靶向IL-18精准治疗炎症性肠病(IBD)的敏感标志物,提出精准治疗IBD的新策略。
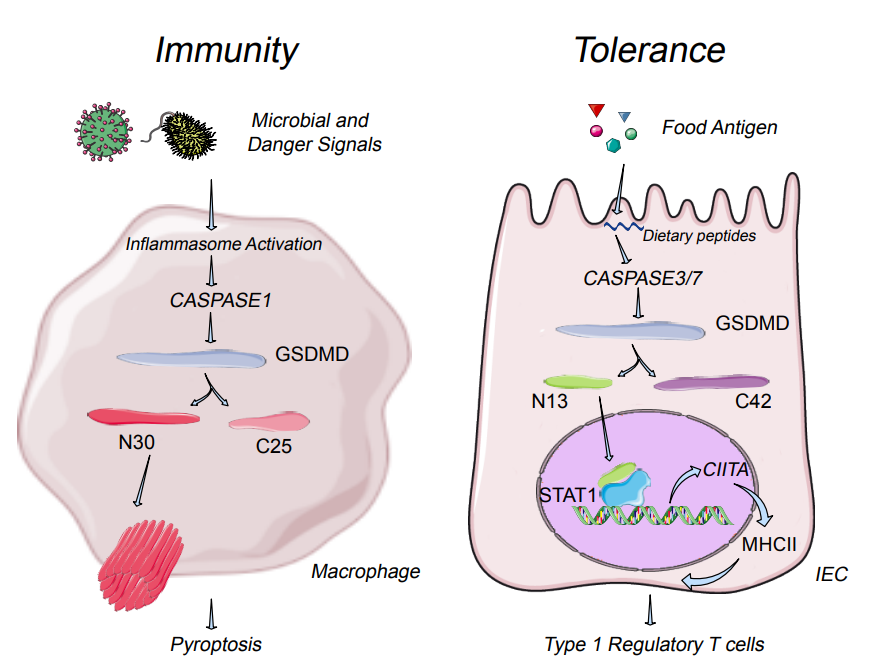
5、揭示食物抗原诱导免疫耐受的机制(CellVolume 186, Issue 14, 6 July 2023, Pages 3033-3048.e20)
朱书教授团队在肠道免疫研究中取得新进展,并对GSDMD在食物耐受中的功能进行了新的解读,该成果于6月15日以题为Gasdermin D licenses MHCII induction to maintain food tolerance in the small intestine的研究论文发表在国际知名期刊《Cell》上。研究人员首先针对生理状态下各个组织细胞的GSDMD进行了蛋白印迹检测,发现检测的各组织样品中只有在小肠的上皮细胞(IEC)中能检测到一条大约13kD大小的剪切带,进一步探索发现这一条剪切带来自GSDMD的N端并且是由食物抗原激活十二指肠IECs中的CASPASE-3/7切割GSDMD的88位天冬氨酸(人源GSDMD是87位)产生的。通过免疫荧光染色以及RNA-seq等实验手段,研究组发现N13片段会入核诱导前端小肠IEC的二类分子表达水平。通过单细胞RNA测序等手段,研究人员发现N13片段缺失造成IEC二类分子下降会进一步导致Tr1细胞减少。由于Tr1细胞也被认为是诱导食物耐受的关键细胞之一,研究人员猜测N13片段最终会参与诱导食物耐受。为此,研究人员针对多种基因表达背景小鼠构建了两组食物耐受模型:花生提取物诱导的花生过敏模型以及OVA诱导的迟发型超敏反应模型,最终证实GSDMD在肠道的生理功能之一是参与构建宿主食物耐受。该工作详细阐述了GSDMD在前端小肠中会在食物诱导下形成一个N13片段,该片段在核孔复合物帮助下入核并辅助增强了STAT1对Ciita的转录调节,使IEC的二类分子表达增加,从而诱导Tr1的上调,最终促进食物耐受形成,为食物过敏的治疗手段提供新思路。
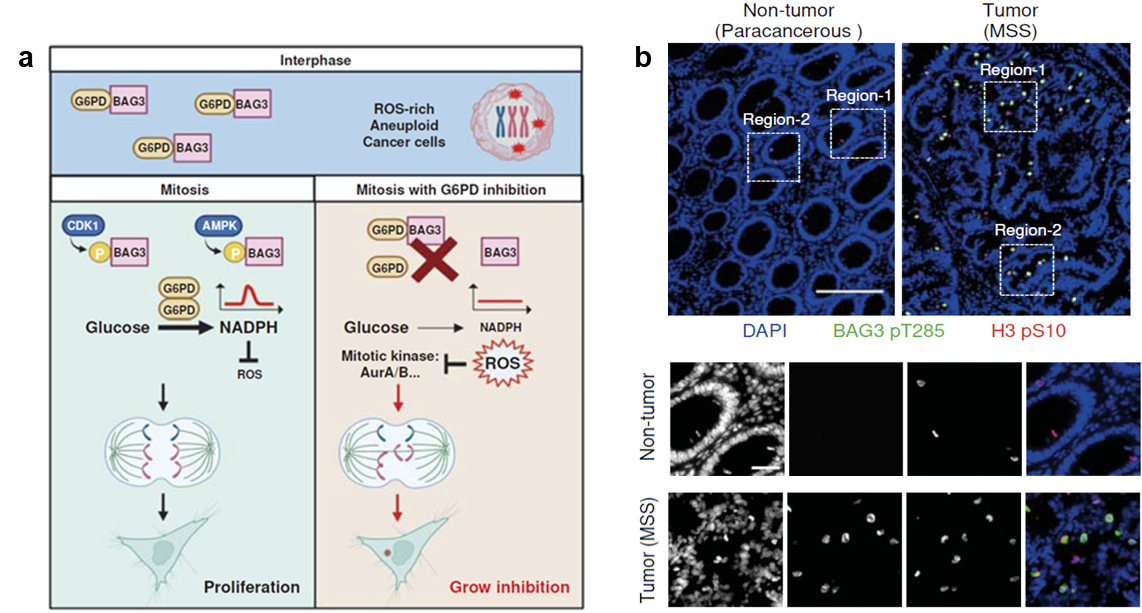
6、揭示NADPH促进非整倍体肿瘤细胞分裂的调控机制(Nature Metabolism volume 5, pages1086–1087 (2023))
2023年6月22日,中国科学技术大学杨振业课题组在Nature Metabolism杂志上发表了题为A mitotic NADPH upsurge promotes chromosome segregation and tumor progression in aneuploid cancer cells的文章,该研究深入地探究了非整倍体肿瘤细胞在分裂期氧化还原状态以及调控机制,发现了核心代谢物NADPH在细胞周期中的变化规律和在有丝分裂期的调控机制,证明了该信号在确保染色体准确分离和维持基因组完整性中的重要功能,从而揭示了非整倍体肿瘤细胞在分裂期特异的代谢调控通路,为临床通过干预代谢通路选择性抑制非整倍体肿瘤提供了新的标记物和治疗新思路。
7、揭示精氨酸甲基化转移酶Prmt1维持精原细胞发育稳态和雄性生育力的机制(Nucleic Acids Research Volume 51, Issue 19, 27 October 2023, 10428–10450)
鲍坚强课题组联合应用多种条件及他莫西芬Tam 诱导敲除Prmt1小鼠动物模型,发现在小鼠精子发生过程中,Prmt1 主要分布在精原细胞核中,且主要调控精原细胞自我更新和分化的发育稳态。结合高通量 CUT&Tag 分析和优化后的微量Smart-seq2 分析,发现Prmt1催化的组蛋白修饰标记 H4R3me2a在启动子和外显子/内含子区域被显著富集,并在小鼠精原细胞中塑造了独特的转录组全貌以及选择性剪接模式。Prmt1敲除显著改变了H4R3me2a与H4R3me2s在全基因组的分布。该成果于2023年9月22日在Nucleic Acids Research杂志在线发表。本项研究揭示了一个核心表观遗传因子Prmt1,在协调高度异质的精原细胞群体自我更新和协调分化过程中的重要功能,包括通过(i)协同调控精原细胞自我更新和分化相关的转录因子的表达;(ii)调控H4R3me2在基因启动子分布来间接调节剪接相关因子的表达;(iii) 通过在外显子/内含子区域不同的H4R3me2 标记来微调可变选择性剪接。这项研究提供了直接的遗传和分子生物学证据,揭示了 Prmt1催化的不对称精氨酸甲基化修饰信号在哺乳动物生殖细胞发育过程中的重要功能。

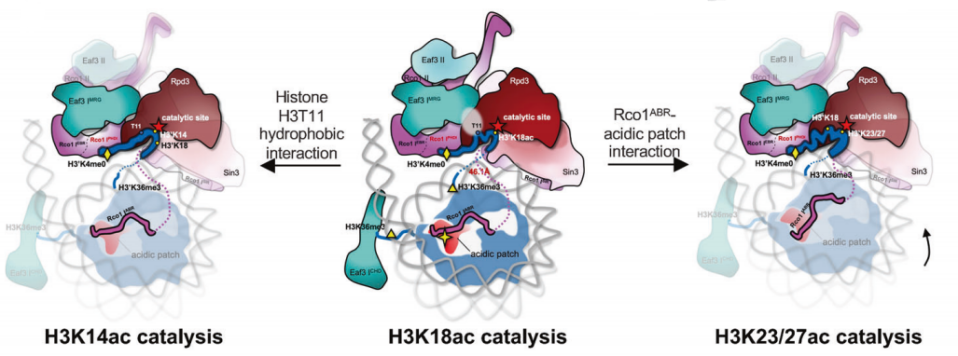
8、揭示Rpd3S/HDAC结合和催化核小体底物的分子机制(Cell Research volume 33, pages971–974 (2023) )
2023年10月16日,中国科学技术大学生命科学与医学部王雪娟、蔡刚教授团队合作在Cell Research(IF=44)期刊上在线发表了题为Structural basis for nucleosome binding and catalysis by the yeast Rpd3S/HDAC holoenzyme的研究论文。该研究利用冷冻电镜技术解析了Rpd3S/HDAC全酶复合物结合H3K36me3修饰核小体底物复合物的高分辨结构,首次清晰捕获了结合在Rpd3S/HDAC活性中心的天然催化底物--组蛋白H3尾部(1-24氨基酸残基),并意外地发现了H3K18残基的侧链对准HDAC催化残基等待催化;并通过功能手段进一步验证了结构上的发现,尤其是Rco1亚基N端结构域在核小体底物结合和活性精准调控中起到的关键作用。
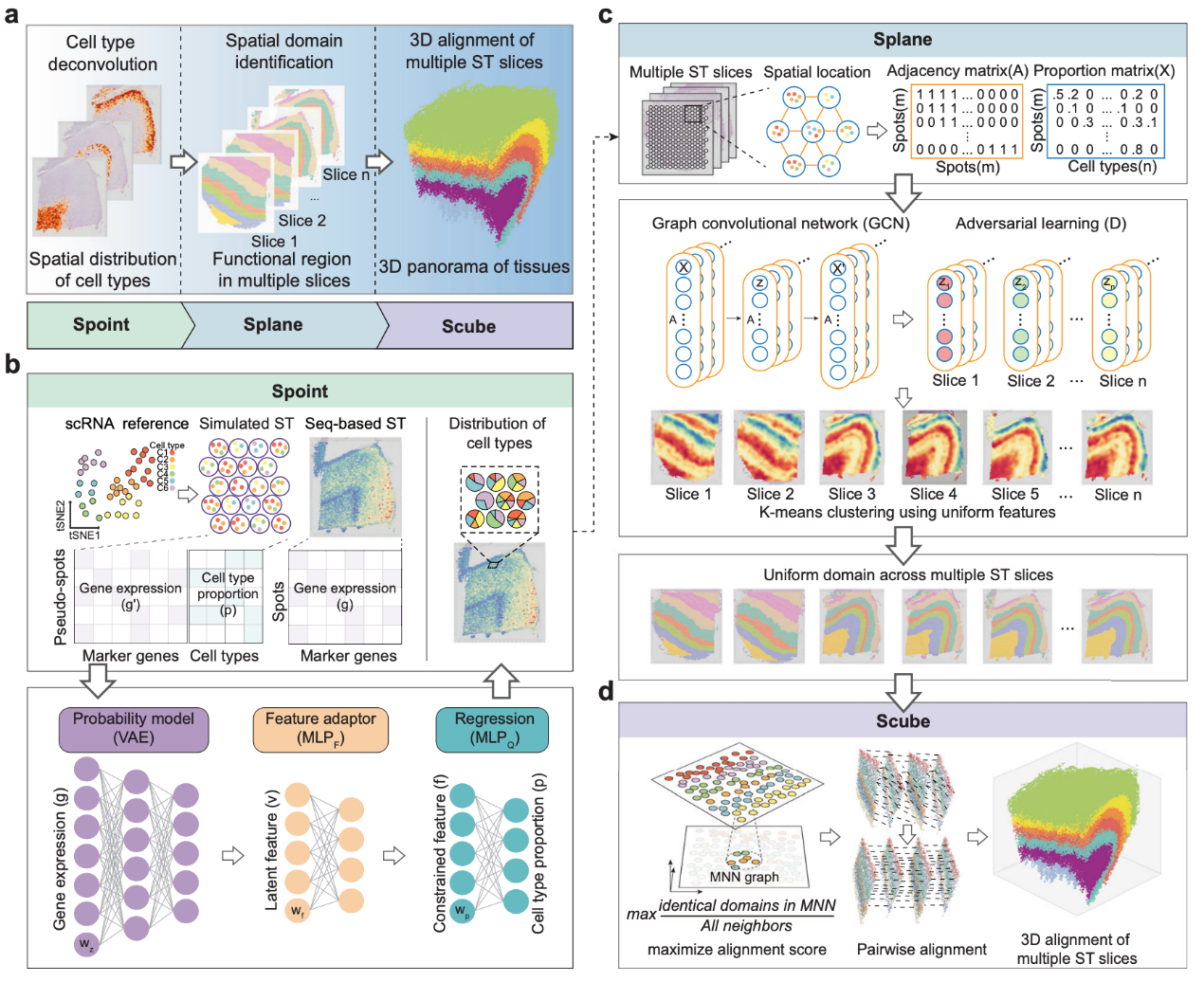
9、开发新型空间转录组分析算法(Nature Communications volume 14, Article number: 7603 (2023) )
中国科学技术大学瞿昆教授课题组成功开发了一种基于深度学习的空间转录组数据分析算法,命名为SPACEL。该算法能够快速高效地处理多个空间转录组切片数据,包括准确预测单个空间点内的细胞类型组成、精准识别跨切片的功能性空间域,以及有效重构复杂组织的三维结构。研究成果以《SPACEL: deep learning-based characterization of spatial transcriptome architectures》为题,发表于2023年11月22日的《自然-通讯》(Nature Communications)杂志。瞿昆教授课题组开发的SPACEL算法包括三个核心模块:Spoint、Splane和Scube,分别对应空间转录组数据分析的三个关键任务。Spoint模块用于预测基于测序的空间转录组数据(如10X Visium)中空间点的细胞类型组成。Spoint采用了单细胞数据模拟的空间点、神经网络模型和统计模型的组合,为估算真实空间转录组数据中的细胞类型比例提供了更稳健和准确的框架。Splane模块则使用细胞类型组成和空间坐标信息,引入了对抗训练到图卷积神经模型中,显著减少了批次效应,从而实现更稳健和高效的空间域识别。对于包含连续切片的空间转录组数据集,Scube模块使用Splane识别的空间域坐标进行对齐,以构建组织的三维结构。Scube采用全局优化策略来实现三维对齐,同时保持整体结构的完整性,使其能够实现更精确的对齐。研究人员将SPACEL应用于11个包括10X Visium、STARmap、MERFISH、Stereo-seq和Spatial Transcriptomics技术的空间转录组数据集,总计156个切片。SPACEL在细胞类型组成预测、空间域识别以及组织三维结构重构等三个核心分析任务上表现出色,明显优于其他同类算法。